Comme dans le cas de nombreux autres organismes végétaux ou animaux, la diversité des poissons est aujourd'hui menacée, à cause de la surexploitation et de la modification des habitats (y compris les pollutions).
De plus, les transferts de patrimoine génétique, intentionnels et accidentels, ont causé des changements génétiques importants dans de nombreuses populations de poissons, principalement dans les eaux douces. Ces impacts ne peuvent être compris que grâce à une connaissance approfondie de la génétique des populations de poissons, en captivité et dans les eaux libres.
L'étude de la génétique a produit une multitude de données comme les caryotypes, les données électrophorétiques, des valeurs d'héritabilité des études sur la sélection et l'amélioration génétique, et des données de génétique moléculaire. Ces données sont largement dispersées dans la littérature ce qui rend les études comparatives très fastidieuses. Les tables de FishBase ont été conçues pour regrouper la majorité de ces données en un ensemble standardisé. Pour permettre l'acquisition, le stockage et l'usage des connaissances sur la génétique, les données ont été divisées en quatre sujets :
Les informations pertinentes pour l'aquaculture sont contenues dans les tables suivantes :
Les chapitres suivants fournissent des détails sur chacune de ces tables.
Christine Casal et Liza Agustin
Les quantités d'ADN nucléaire et cellulaire (voir Fig. 50) sont importantes pour les études de génétique et de systématique des poissons.
Les champs
Chromosome number [Nombre de chromosomes] : Plusieurs champs indiquent le nombre de chromosomes haploïde/gamétique et diploïde/zygotique. Si ce nombre est variable, l'intervalle est indiqué pour le nombre diploïde.

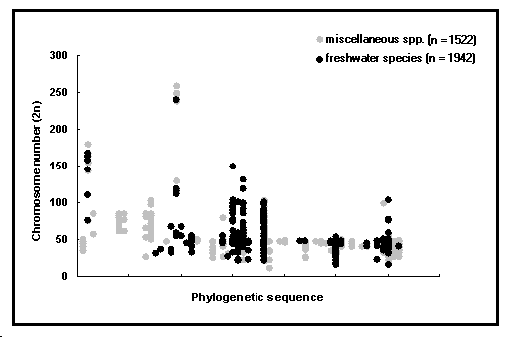
Fig. 50. Nombre de chromosomes des poissons d'eau douce comparé à celui de diverses espèces ordonnées en une séquence phylogénétique, des groupes les plus anciens (gauche) aux plus récents (droite). Noter la diminution de ce nombre et la variance pour les groupes récents. Voir Encadré 29 pour une interprétation de ce graphique.
Chromosome types [Types de chromosome] : Plusieurs champs indiquent le nombre de chromosomes de chacun des types suivants :
Chromosome arm number [Nombre de bras des chromosomes] : Indique le nombre total des bras des chromosomes qui dépend en grande partie du type de chromosomes (par exemple, un chromosome métacentrique aura deux bras alors qu'un chromosome télocentrique n'en aura qu'un).
Sex-determining mechanism [Déterminisme du sexe] : Indique le type de déterminisme du sexe mâle-femelle parmi les choix suivants : xx-xy ; xx-xo ; etc. pour les espèces à chromosome sexuel ; ou aucun chromosome hétéromorphe lié au sexe.
Genetic marker(s) [Marqueur(s) génétique(s)] : Indique si un(des) marqueur(s) génétique(s) existe(nt) pour l'espèce par oui/non. Un marqueur est un trait phénotypique (par exemple un allozyme, une bande chromosomique, etc.) qui peut permettre d'inférer le génotype.
DNA content [Contenu ADN] : Indique le contenu cellulaire haploïde spécifique (en pg). Si des références proposent des valeurs différentes de celles saisies dans ce champ, elles sont signalées dans les Remarques.
DNA sequencing [Séquençage ADN] : Indique si des séquençages d'ADN ont été réalisés pour cette espèce.
mtDNA analysis [Analyse ADNmt] : Indique si des études sur l'ADN mitochondrial ont été réalisées pour cette espèce.
Remarks [Remarques] : Contient par exemple tout commentaire sur la présence de nouveaux arrangements structurels, de caractéristiques chromatiques spécialisées, ou de déterminisme du sexe particulier, de phénomène de polyploïdisation.
| Encadré 29. ADN, taille cellulaire et nage
chez les poissons. Le contenu d'ADN cellulaire
chez les plantes et les animaux est extrêmement
variable. Peu de généralisations ont été établies
qui permettent de prédire la quantité d'ADN dans les
cellules d'un groupe d'organismes donné. Références |
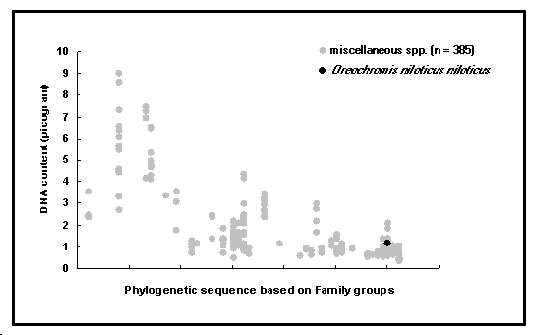
Fig. 51. Contenu en ADN cellulaire chez Oreochromis niloticus niloticus. La diminution du contenu d'ADN des groupes anciens (gauche) aux groupes récents (droit) est semblable à la diminution du nombre de chromosomes (Fig. 50).
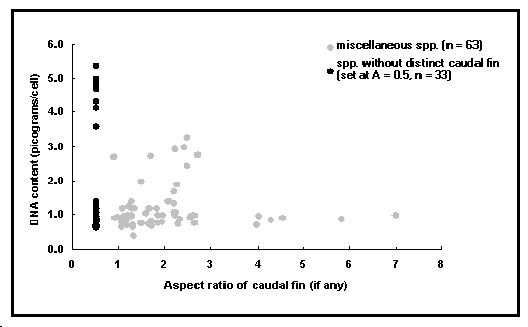
Fig. 52. Contenu d'ADN cellulaire, pris ici comme une mesure de taille cellulaire en fonction de l'indice de forme de la nageoire caudale (A), prise ici comme mesure d'activité. Voir Encadré 29 pour une discussion de ce graphique, et Fig. 46 pour la définition de l'indice de forme de la nageoire caudale.
Statut
La table GENETICS contient des informations sur plus de 1 500 espèces extraites de plus de 350 références.
Sources
Nous avons utilisé des compilations de nombres de chromosomes et de caryotypes de différents groupes de poissons, entre autres Post (1965), Hinegardner et Rosen (1972), Gold et al. (1980) Agnèse et al. (1990), Gold et al. (1990), Jianxun et al. (1991), Porto et al. (1992), Suzuki (1992) et Vasil'yev et Grogoryan (1992). Mais la majorité de nos sources sont des articles qui ne traitent que d'une seule à quatre espèces, comme Fontana (1994).
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Genetics dans la vue BIOLOGY, et sur le bouton Genetics dans la fenêtre GENETICS.
Remerciements
Nous remercions le Monsieur P. Yershov pour ses conseils sur la structure et le contenu de cette table.
Références
Agnèse, J.-F., T. Oberdorff et C. Ozouf-Costaz. 1990. Karyotypic study of some species of family Mochokidae (Pisces, Siluriformes) : evidence of female heterogamety. J. Fish Biol. 37 : 375-381.
Fontana, F. 1994. Chromosomal nucleolar organizer regions in four sturgeon species as markers of karyotype evolution in Acipenseriformes (Pisces). Genome 37(5) : 888-892.
Gold, J.R., W.J. Karel et M.R. Strand. 1980. Chromosome formulae of North American fishes. Prog. Fish Cult. 42 : 10-23.
Gold, J.R., C.J. Ragland et L.J. Schliesing. 1990. Genome size variation and evolution in North American cyprinid fishes. Genet. Sel. Evol. 22 : 11-29.
Hinegardner, R. et D.E. Rosen. 1972. Cellular DNA content and the evolution of teleostean fishes. Am. Nat. 106(951) : 621-644.
Jianxun, C., R. Xiuhai et Y. Qixing. 1991. Nuclear DNA content variation in fishes. Cytologia 56 : 425-429.
Porto, J.I.R., E. Feldberg, C.M. Nakayama et J.N. Falcao. 1992. A checklist of chromosome numbers and karyotypes of Amazonian freshwater fishes. Rev. Hydrobiol. Trop. 25(4) : 287-299.
Post, A. 1965. Vergleichende Untersuchungen der
Chromosomenzahlen bei Sü![]() wasser-Teleosteern.
Z. Zool. Syst. Evolut. Forsch. 3 : 47-93.
wasser-Teleosteern.
Z. Zool. Syst. Evolut. Forsch. 3 : 47-93.
Suzuki, A. 1992. Chromosome and DNA studies of eight species in the family Cobitidae (Pisces, Cypriniformes). Kromosome 67-68 : 2275-2282.
Vasil'yev, V.P. et K.A. Grogoryan. 1992. Karyology of fishes from the family Gobiidae. Vopr. Ikhtiol. 32(5) : 27-40.
Christine Casal et Liza Agustin
Les informations déduites des études électrophorétiques ont été structurées en trois tables : la table ELECSTUDIES contient des informations générales par espèce sur des études qui ont été conduites sur différentes populations de cette espèce ; la table ELECDAT contient les données par locus et par étude ; et la table ELECSUB contient les allèles qui ont été détectés par locus.
Ensemble, ces tables fournissent des informations sur la structure génétique et la variabilité des populations de poissons sauvages et en élevage. Ces informations sont importantes pour la sélection des espèces ou des souches pour l'aquaculture. Elles faciliteront les programmes de gestion et de conservation des stocks naturels.
Au fur et à mesure que de nouvelles données étaient saisies dans cette table, il est devenu possible de mettre en évidence les lacunes actuelles dans les connaissances (c'est-à-dire les espèces importantes encore peu étudiées), ainsi que les méthodes et les formats pour créer les rapports dans FishBase les plus appropriés pour la caractérisation génétique des diverses espèces.
Les tables contiennent des fréquences alléliques d'études électrophorétiques de populations sauvages et en élevage. Elles contiennent aussi des informations sur les enzymes, le nombre total de locus étudiés, les tissus et les systèmes de tampon utilisés, des valeurs d'hétérozygotie et des proportions de locus polymorphes.
Les champs
Locality and Country [Localité et Pays] : Indique le lieu de capture des spécimens.
Total loci [Nombre total de locus] : Indique le nombre total de locus examinés.
Observed heterozygosity [Hétérozygotie observée] : Indique la proportion d'individus hétérozygotes dans une population pour un nombre donné de locus. Un individu est dit homozygote s'il possède deux allèles identiques au même locus, et hétérozygote s'ils sont différents.
Expected heterozygosity [Hétérozygotie attendue] : Indique la proportion d'individus hétérozygotes qui est calculée à partir de fréquences alléliques connues en supposant que la population est à l'équilibre de Hardy-Weinberg. Elles sont calculés par locus, population et espèce, et permettent d'apprécier le potentiel pour un élevage sélectif (voir Fig. 53).
Polymorphic loci [Locus polymorphes] : Indique le nombre de locus polymorphes dans un échantillon rapporté au nombre total de locus examinés (voir Fig. 54). Pour standardiser les données, le critère de 95 % est appliqué ici, par lequel un locus est considéré polymorphe si la fréquence allélique la plus forte ne dépasse pas 0,95. Si le critère de 99 % a été appliqué, une indication est notée dans Comment [Commentaire].
Enzyme : Indique les noms, les abréviations et les codes numériques standardisés pour les enzymes et autres protéines communément analysées dans les travaux de génétique sur les poissons. Les noms et les codes utilisés sont ceux de la nomenclature recommandée par le comité de l'International Union of Biochemistry (Shaklee et al. 1990).
Locus : Indique la position spécifique ou l'emplacement d'un gène sur le chromosome. Un gène est une longueur spécifique d'ADN (acide désoxyribonucléique) occupant un locus. Un locus est appelé monomorphe si un seul allèle du gène situé à ce locus est connu, et polymorphe quand plusieurs allèles sont connus. Si deux locus ou plus sont impliqués dans la production de formes différentes d'une protéine (isozymes), le locus le plus anodique est numéroté 1, le suivant 2, etc. Quelquefois, le locus est désigné par les lettres, le plus anodique est indiqué A, le suivant B, etc.
Tissue : Le type du tissu échantillonné pour l'électrophorèse parmi les choix suivants : skeletal muscle [muscle squelettique] ; visceral muscle [muscle viscéral] ; heart [cœur] ; kidney [rein] ; liver [foie] ; blood [sang] ; mucus [mucus] ; eye lens [cristallin] ; whole body [corps entier] ; others [autres]. Le choix est précisé dans Comment [Commentaire].
Method used [Méthode utilisée] : Indique le type de méthode d'électrophorèse utilisé parmi les choix suivants : starch gel [gel d'amidon] ; polyacrylamide gel [gel de polyacrylamide ] ; sodium dodecyl sulfate [dodecyl-sulfate de sodium] ; other methods [autres méthodes]. L'électrophorèse sur gel est une des méthodes les plus communes pour étudier la variation génétique des individus au niveau de la souche et de l'espèce.
Buffer system [Tampon] : Indique le tampon utilisé pendant l'électrophorèse pour la séparation claire des protéines et des enzymes spécifiques. Les 15 tampons les plus communément utilisés sont décrits par Boyer et al. (1963), Ridgway et al. (1970), Shaw et Prasad (1970), Selander et al. (1971), et Clayton et Tretiak (1972).
pH : Indique le pH du tampon utilisé.
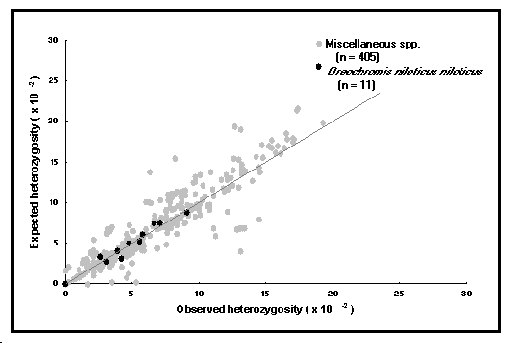
Fig. 53. Hétérozygotie prédite en fonction de l'hétérozygotie observée chez Oreochromis niloticus niloticus (points noirs) et chez divers poissons (points blancs). La diagonale a une pente de 1, et identifie les points où l'hétérozygotie prédite est égale à celle observée. Les valeurs largement au-dessus peuvent résulter de la consanguinité, celles largement au-dessous, du croisement de souches.
Samples [Échantillons] : Indique le nombre des spécimens par localité ou par population étudiée.
Allele [Allèle] : Indique l'un des allèles (c'est-à-dire l'une des formes alternatives) du gène étudié. Les allèles sont distingués pendant l'électrophorèse par leurs produits protéiniques (enzymes). La mobilité électrophorétique relative des enzymes dans un zymogramme est exprimée en nombres. Les mobilités relatives sont calculées en assignant 100 à l'allèle anodique le plus commun (ou -100 pour un locus cathodique). Le signe négatif est attribué aux allèles à mobilité cathodique.
Allele frequency [Fréquence allélique] : Elle est calculée à un locus donné en utilisant la formule suivante : fréquence allélique A = 2 . (fréquence génotypique AA) + (fréquence génotypique Aa) / 2n, où n = nombre d'individus étudiés.
Statut
Les tables ELECSTUDIES, ELECDAT et ELECSUB contiennent actuellement plus de 11 000 enregistrements (chaque enregistrement traite les allèles d'un seul locus) de fréquences alléliques pour plus de 900 études et plus de 800 populations/souches de poissons. La mise à jour de ces tables avec l'aide de D.O.F. Skibinski et ses collaborateurs, à partir des références qu'ils ont signalées (Skibinski et al. 1991), en font la plus grande compilation de la variabilité génétique des poissons.
Graphiques
Plusieurs graphiques peuvent être affichés à partir de cette table :
Tous ces graphiques peuvent être affichés à partir de la vue GENETICS et mettent en valeur l'espèce sélectionnée. Ou bien, vous pouvez les afficher à partir de la fenêtre REPORTS puis GRAPHS.
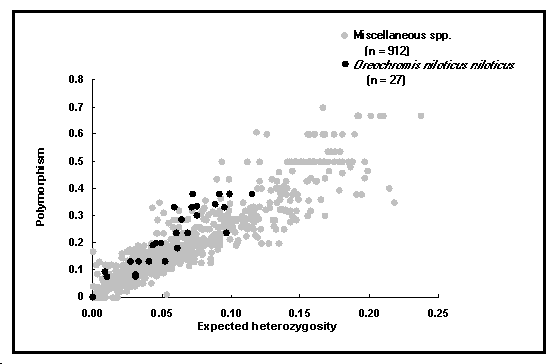
Fig. 54. Polymorphie en fonction de l'hétérozygotie prédite pour Oreochromis niloticus niloticus (points noirs) et diverses espèces (points ouverts) ; un graphique semblable, accessible du menu Graphs, peut être utilisé pour comparer les populations en captivité avec celles en eaux libres. Le graphique montre que le tilapia du Nil est plus polymorphe que les autres espèces comparées.
Sources
Les références majeures déjà utilisées sont Winans (1980), McAndrew et Majumdar (1983), Macaranas et al. (1986, 1995), van der Bank et al. (1989), Carvalho et al. (1991) et Pouyaud et Agnèse (1995).
Compiler entièrement les fréquences alléliques et les informations afférentes déjà publiées est un défi plutôt redoutable. Il implique la résolution de problèmes dus au manque de standardisation entre les publications qui empêche toujours le regroupement des données (Agustin et al. 1993, 1994).
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Genetics dans la vue BIOLOGY et sur le bouton Allele frequencies dans la fenêtre GENETICS. Double-cliquer sur une des lignes dans la vue LIST OF AVAILABLE ELECTROPHORETIC STUDIES affiche les informations relatives à l'étude désignée.
Cliquer sur le bouton Electrophoretic data dans la vue INFORMATION ON HETEROZYGOTY AND POLYMORPHISM affiche la liste des locus (enzymes). Double-cliquer sur une des lignes affiche les informations sur le locus désigné.
Cliquer sur le bouton Allele Frequencies dans la vue LOCUS INFORMATION affiche les fréquences alléliques (‹ La table ELECDAT ›)
Remerciements
Nous remercions R.E. Brummett, A.E. Eknath, G.C. Mair, J.G. McGlade, D. Pauly, R.S.V. Pullin et D.O. Skibinski pour leur conseil sur la structure et le contenu de cette table.
Références
Agustin, L.Q., R. Froese, A.E. Eknath et R.S.V. Pullin. 1993. Documentation of genetic resources for aquaculture - the role of FishBase, p. 63-68. In D. Penman, N. Roongratri et B. McAndrew (éds). International Workshop on Genetics in Aquaculture and Fisheries Management. ASEAN-EEC Aquaculture Development and Coordination Programme, Bangkok, Thailand.
Agustin, L.Q., M.L.D. Palomares et G.C. Mair. 1994. FishBase : a repository of genetic information on fish. Poster presented at the Fifth International Symposium on Genetics in Aquaculture, 19-25 June 1994, Dalhousie University, Halifax, Nova Scotia, Canada.
Boyer, S.H., D.C. Fainer et E.J. Watson-Williams. 1963. Lactate dehydrogenase variant from human blood : evidence for molecular subunits. Science 141 : 642-643.
Carvalho, G.R., P.W. Shaw, A.E. Magurran et B.H. Seghers. 1991. Marked genetic divergence revealed by allozymes among populations of the guppy Poecilia reticulata (Poeciliidae), in Trinidad. Biol. J. Linn. Soc. 42 : 389-405.
Clayton, J.W. et D.N. Tretiak. 1972. Amine-citrate buffers for pH control in starch gel electrophoresis. J. Fish. Res. Board Can. 29 : 1169-1172.
Macaranas, J.M., N. Taniguchi, M.J.R. Pante, J.B. Capili et R.S.V. Pullin. 1986. Electrophoretic evidence for extensive hybrid gene introgression into commercial Oreochromis niloticus (L.) stocks in the Philippines. Aquacult. Fish. Manage. 17 : 249-258.
Macaranas, J.M., L.Q. Agustin, M.C.A. Ablan, M.J.R. Pante, A.E. Eknath et R.S.V. Pullin. 1995. Genetic improvement of farmed tilapias : biochemical characterization of strain differences in Oreochromis niloticus. Aquaculture International 3 : 43-54.
McAndrew, B.J. et K.C. Majumdar. 1983. Tilapia stock identification using electrophoretic markers. Aquaculture 30 : 249-261.
Nelson, J.S. 1994. Fishes of the world. 3ème édition. John Wiley and Sons, New York. 600 p.
Pouyaud, L. et J.-F. Agnèse. 1995. Phylogenetic relationships between 21 species of three tilapiine genera Tilapia, Sarotherodon and Oreochromis using allozyme data. J. Fish Biol. 47(1) : 26-38.
Ridgway, G.J., S.W. Sherburne et R.D. Lewis. 1970. Polymorphism in the esterases of Atlantic herring. Trans. Am. Fish. Soc. 99 : 147-151.
Selander, R.K., M.H. Smith, S.Y. Yang, W.E. Johnson et J.B. Gentry. 1971. Biochemical polymorphism and systematics in the genus Peromyscus. I. Variation in the old field mouse (Peromyscus polionotus). Studies in Genetics VI. Univ. Texas Publ. 7103 : 49-90.
Shaklee, J.B., F.W. Allendorf, D.C. Morizot et G.S. Whitt. 1990. Gene nomenclature for protein-coding loci in fish. Trans. Am. Fish. Soc. 119 : 2-15.
Shaw, C.R. et R. Prasad. 1970. Starch gel electrophoresis of enzymes - a compilation of recipes. Biochem. Genet. 4 : 297-320.
Skibinski, D.O.F., M. Woodwark et R.D. Ward. 1991. The protein diversity database. University College of Swansea, Singleton Park, Swansea, Wales and CSIRO Division of Fisheries, Tasmania, Australia. 16 p.
van der Bank, F.H., W.S. Grant et J.T. Ferreira. 1989. Electrophoretically detectable genetic data for fifteen southern African cichlids. J. Fish Biol. 34 : 465-483.
Winans, G.A. 1980. Geographic variation in the milkfish Chanos chanos. I. Biochemical evidence. Evolution 34(3) : 558-574.
Christine Casal et Liza Agustin
L'objectif de cette table est de faciliter l'application des concepts et des méthodes de la génétique à l'aquaculture moderne. Elle contient ainsi des enregistrements d'héritabilité et de réponses à la sélection. L'amélioration génétique des poissons d'élevage exige des programmes pour sélectionner les traits d'importance économique élevée (comme le taux de croissance, l'âge à maturité, la qualité de la carcasse et bien d'autres ; voir Encadré 30).
Les champs
Locality and Country [Localité et le pays] : Indique le lieu où l'expérience a été effectuée.
Trait [Trait] : Indique le trait phénotypique désiré pour l'amélioration par sélection, parmi les choix suivants : growth rate [taux de croissance] ; age at first maturity [âge à maturité] ; size at first maturity [taille à maturité] ; egg number [nombre d'œufs] ; egg size [taille des œufs] ; egg weight [poids des œufs] ; egg survival [survie des œufs] ; larval survival [survie des larves] ; disease resistance [résistance aux maladies] ; behavior [comportement] ; resistance to environmental factors [résistance aux facteurs environnementaux] ; dressing weight [poids éviscéré] ; carcass quality [qualité de la carcasse] ; fat content [contenu en lipides] ; protein content [contenu en protéines] ; food conversion [conversion de la nourriture] ; anatomical modification color and other [couleur de la modification anatomique et autre]. Les traits qui ne sont pas listés sont précisés dans le premier champ Comment [Commentaire].
Mean [Moyenne] : Indique la valeur moyenne du trait étudié.
Unit [Unité] : Indique l'unité de mesure du trait (par exemple, g, semaines, mm).
S.D. [Écart type] : Indique l'écart-type de l'héritabilité.
C.V. [C.V.] : Coefficient de variation du trait examiné, défini par la formule C.V. = écart-type/moyenne.
Heritability (h2) [Héritabilité (h2)] : Indique le pourcentage de variance génétique additive dans la variation phénotypique totale, si le trait sera exprimé ou sera passé à la progéniture. Si un trait est suffisamment héritable, il est possible que l'élevage par sélection soit très efficace. Cependant, si h2 est faible, les facteurs environnementaux sont la cause de la majeure partie de la variation et par conséquent, tout gain génétique obtenu par sélection sera limité voire nul.
S.E. [Erreur-standard] : Indique l'erreur-standard de la moyenne d'héritabilité.
Method [Méthode] : Indique la méthode utilisée pour l'estimation de l'héritabilité parmi les choix suivants : sib analysis [évaluation sur collatéraux] ; offspring/parent regression [régression progéniture/parent] ; realized heritability [héritabilité réalisée] ; others [autres]. Les méthodes qui ne sont pas listées sont précisées dans le second champ Comment [Commentaire].
Selection studies [les études sur la sélection] : Indique si une étude de sélection a été réalisée.
Response % [% Réponse] : Indique la réponse à la sélection exprimée en pourcentage.
Method [Méthode] : Indique la méthode de sélection parmi les choix suivants : mass selection [sélection collective] ; individual selection [sélection individuelle] ; sib selection [sélection sur collatéraux] ; family selection [sélection familiale] ; within family selection [sélection intra-familiale] ; index selection and tandem selection [indice de sélection et tandem de sélection] ; others [autres]. Les méthodes qui ne sont pas listées sont précisées dans le troisième champ Comment [Commentaire].
Statut
La table GENEDAT contient 200 enregistrements pour 9 espèces et souches, extraits entre autres de Gjedrem (1983), Gjerde (1986) et Tave (1988).
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Genetics dans la vue BIOLOGY et sur le bouton Heritability dans la fenêtre GENETICS.
Références
Gjedrem, T. 1983. Genetic variation in quantitative traits and selective breeding in fish and shellfish. Aquaculture 33 : 51-72.
Gjerde, B. 1986. Growth and reproduction in fish and shellfish. Aquaculture 57 : 37-55.
Tave, D. 1988. Genetics and breeding of tilapia : a review, p. 285-293. In R.S.V. Pullin, T. Bhukaswan, K. Tonguthai et J.L. Maclean (éds) The Second International Symposium on Tilapia in Aquaculture. ICLARM Conf. Proc. 15, 623 p.
Christine Casal et Liza Agustin
Cette table documente l'ascendance des souches cultivées de poissons. Elle a été développée à l'origine pour servir de registre des souches de tilapia recommandé pendant The Second International Symposium on Tilapia in Aquaculture (ISTA II) en 1987, à Bangkok, Thaïlande (Pullin 1988). Les informations ont par la suite été étendues pour intégrer d'autres espèces utilisées en aquaculture (voir Encadré 30), comme recommandé dans les Articles 7 et 10 de la Convention sur la Diversité Biologique (UNEP 1992). Les données génétiques, y compris l'histoire de la population fondatrice, la gestion du stock de géniteurs, le statut de la souche et la description des caractères distinctifs des souches faciliteront l'utilisation et la conservation de la variabilité génétique intraspécifique dans l'aquaculture. La standardisation de la nomenclature reste un problème et pour l'instant, aucun système n'est universellement accepté pour dénommer des souches dans une espèce.
Comme la domestication des espèces aquatiques progresse, on peut s'attendre à ce que des souches génétiquement plus distinctes apparaissent. Ces souches peuvent émerger par le simple fait de la domestication, accompagnée d'un flux génique restreint entre les fermes, ou par des pratiques de sélection, comme l'élevage par sélection, les manipulations chromosomiques (polyploïdisation et inversion sexuelle), le transfert de gène et/ou l'hybridation.
Le registre officiel des souches peut servir de source pour localiser des poissons avec des traits spécifiques, par exemple, des tilapias de couleur rouge, et pour suivre les technologies d'amélioration génétique. Cependant, le registre peut servir aussi de liste de surveillance pour les souches potentiellement menacées, en enregistrant le nombre d'individus reproducteurs dans la souche, semblable au World Watch List for Domestic Animal Diversity [Liste de surveillance mondiale pour la diversité des animaux domestiques] (Scherf 1995), où les races/souches en danger peuvent être enregistrées et recommandées pour des efforts immédiats de conservation.
Un résumé de la description des souches d'élevage est contenu dans le champ Stock definition [Définition du stock] de la table STOCKS. Il indique le nom de la souche, son année originale de transfert et la taille du stock originel.
Les champs
Country [Pays] : Indique le pays où la souche est trouvée.
StrainCode [Code souche] : Combinaison unique de lettres et d'un nombre à 3 chiffres. Les lettres 1-2 sont les deux premières lettres du genre ; les lettres 3-5 sont les trois premières lettres de l'épithète spécifique (espèce) ; les lettres 6-7 sont les deux premières lettres de l'épithète subspécifique (sous-espèce). Le nombre est un numéro d'ordre. Si l'origine de la souche n'est pas une sous-espèce, les lettres 6-7 sont XX. Pour les hybrides, les lettres 6-7 sont HX.
Trait [Trait] : Indique le(s) trait(s) qui distingue(nt) une souche de son stock fondateur parmi des choix identiques à ceux de la table GENEDAT ci-dessus.
Size of founding stock [Taille du stock fondateur] : Indique l'effectif des membres fondateurs composant la population originelle.
Breeding strategy [Stratégie de reproduction] : Indique la méthode de reproduction dans l'élevage parmi les choix suivants : manipulation chromosomique (polyploïdisation et inversion sexuelle) ; élevage de sélection ; hybridation ; transfert de gènes ; accouplement normal.
Is strain reproducing (Y/N) ? [La souche se reproduit-elle (O/N) ?] : Indique si la souche est reproductivement viable. Par exemple, une souche ne comprenant que des femelles de truites triploïdes ne serait pas capable de se reproduire.
Female [Femelle] : Indique l'effectif de femelles fondatrices dans la population originelle.
Male [Mâle] : Indique l'effectif de mâles fondateurs dans la population originelle.
Number of broodstock [Nombre de géniteurs] : Indique le nombre actuel de géniteurs, champ qui facilite la détermination du statut de conservation et de menace d'extinction de la souche.
Year of first breeding [Année de première reproduction] : Fait référence à l'année où le stock fondateur s'est reproduit pour la première fois.
Source of founding stock [Origine du stock fondateur] : Indique la provenance du stock originel. Le pays est aussi indiqué.
StrainCode of source [Code de la souche originelle] : Indique le code du stock d'où proviennent les spécimens fondateurs.
Year of arrival [année d'arrivée] : Indique l'année d'arrivée du stock originel sur son nouvel emplacement ou dans sa nouvelle localité.
Availability of strain [Disponibilité de la souche] : Indique où la souche est utilisée et comment elle peut être acquise.
Statut
La table STRAINS, ou registre des souches, est seulement préliminaire : ses enregistrements n'ont pas été vérifiés et elle inclut seulement un peu plus de 70 souches de tilapias (Oreochromis spp.), de carpe commune (Cyprinus carpio carpio) et d'une espèce de labéo (Labeo rohita).
Sources
Les références majeures déjà utilisées sont Khater et Smitherman (1988), Pullin (1988), Pullin et Capili (1988), Komen (1990) et Eknath et al. (1993).
Nous avons prévu d'intégrer plus d'hybrides et des souches génétiquement améliorées, en suivant la structure du registre des souches de truites (National Trout Strain Registry) de Kincaid et Brimm (1994).
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Genetics dans la vue BIOLOGY, et sur le bouton Strains dans la fenêtre GENETICS.
Les souches étant enregistrées pour l'espèce à laquelle elles appartiennent, cliquer sur le bouton Biology dans la vue SPECIES affiche une liste comprenant une ligne pour l'espèce en général et une ligne par souche d'élevage. Double-cliquer sur une des lignes de cette liste affiche la vue BIOLOGY correspondante. La description de cette procédure n'a pas été incluse dans les chapitres équivalents des autres tables, mais est implicite pour toutes celles qui sont accessibles par le bouton Biology de la vue SPECIES.
Remerciements
Nous remercions Mlle Liza Agustin, Ambekar E. Eknath, Harold Kincaid, Wolfgang Villwock et Ulricke Sienknecht pour leur conseil sur la structure et le contenu de cette table. Nous remercions Harold Kincaid pour nous avoir offert une copie du logiciel gérant le National Trout Strain Registry.
Références
Eknath, A.E., M.M. Tayamen, M.P. de Vera, J.C. Danting, R.A. Reyes, E.E. Dionisio, J.B. Capili, H.L. Bolivar, T.A. Abella, A.V. Circa, H.B. Bentsen, B. Gjerde, T. Gjedrem et R.S.V. Pullin. 1993. Genetic improvement of farmed tilapias : the growth performance of eight strains of Oreochromis niloticus tested in different farm environments. Aquaculture 111 : 171-188.
Khater, A. A. et R.O. Smitherman. 1988. Cold tolerance and growth of three strains of Oreochromis niloticus, p. 215-218. In R.S.V. Pullin, T. Bhukaswan, K. Tonguthai et J.L. Maclean (éds) The Second International Symposium on Tilapia in Aquaculture. ICLARM Conf. Proc. 15, 623 p.
Kincaid, H. et S. Brimm. 1994. National Trout Strain Registry. U.S. Fish and Wildlife Service's Division of Fish Hatcheries, National Fishery Research and Development Laboratory and Office of Administration - Fisheries, USA.
Komen, J. 1990. Clones of common carp, Cyprinus carpio : new perspectives in fish research. Agricultural University Wageningen, Wageningen, Netherlands. Thèse de Doctorat. 169 p.
Pullin, R.S.V., Éditeur. 1988. Tilapia genetic resources for aquaculture. ICLARM Conf. Proc. 16, 108 p.
Pullin, R.S.V. et J.B. Capili. 1988. Genetic improvement of tilapias : problems and prospects, p. 259-266. In R.S.V. Pullin, T. Bhukaswan, K. Tonguthai et J.L. Maclean (éds) The Second International Symposium on Tilapia in Aquaculture. ICLARM Conf. Proc. 15, 623 p.
Scherf, B.D. 1995. World Watch List for Domestic Animal Diversity. Food and Agriculture Organization of the United Nations, Rome, Italy. 769 p.
UNEP. 1992. Convention on Biological Diversity. United Nations Environment Programme, Nairobi, Kenya. 52 p.
Christine Casal et Devin Bartley
La connaissance des performances d'élevage des poissons selon plusieurs systèmes d'aquaculture est utile pour évaluer les potentialités d'une espèce pour l'aquaculture et pour aider au choix de méthodes d'aquaculture et de systèmes de production appropriés à cette espèce.
Le but principal de la table CULTSYS est de résumer les données sur les expériences en aquaculture. La table contient des informations sur les systèmes expérimentaux et leurs paramètres physico-chimiques, la qualité et la quantité des intrants nutritifs et la production, par espèce (voir aussi ‹ La table CULTSPEC ›, ce volume). Elle constitue un modèle que les scientifiques peuvent suivre pour documenter des expériences d'aquaculture.
Les champs
Name [Nom] : Indique le nom de la ferme, de la station ou de l'institut de la réalisation de l'expérimentation, complété parfois avec les Latitude, Longitude et Altitude.
Year [Année] : Indique l'année de l'expérimentation.
Type of culture [Type de élevage] : Indique si une ou plusieurs espèces sont élevées simultanément (mono- ou polyculture).
Sex [Sexe] : Indique une possible ségrégation sexuelle parmi une liste de procédures utilisées en aquaculture : monosex male [mâle monosexe] ; monosex female [femelle monosexe] ; mixed sex [mixte].
Culture system I [Système d'élevage I] : Indique une classification générale du système de production parmi les choix suivants : intensive (high density, food added) [intensif (forte densité d'individus, adjonction de nourriture)] ; semi-intensive (medium density, no food but fertilizer added) [semi-intensif (densité moyenne, pas d'adjonction de nourriture, mais épandage d'engrais)] ; extensive (low density, nothing added) [extensif (faible densité, aucune adjonction)] ; experimental [expérimental].
Culture system II [Système d'élevage II] : Précise le système de production parmi les choix : ponds [étangs] ; integrated farm pond system [système d'étang fermier intégré] ; sewage - (excreta and wastewater) fed system [système à épandage d'eaux usées] ; rice field [rizière] ; raceways [bassins à eau courante] ; static tanks [réservoirs statiques] ; silos [silos] ; cages [cages] ; pens [enclos] ; farm dams [retenues artificielles] ; other (see Description of culture system) [autre (voir le champ Description of culture system [description du système d'élevage])].
Production units [unité de production] : Indique le nombre d'unités de production, la superficie Area (ha), la profondeur moyenne Average depth (m) et le volume Volume des unités expérimentales (m3).
Main water source [Source principale d'eau] : Parmi les choix : rainfall [pluie] ; spring [source] ; river/creek [rivière/ruisseau] ; lake [lac] ; reservoir [réservoir] ; estuary [estuaire] ; lagoon [lagune] ; ocean [océan] ; groundwater [eau souterraine] ; tap water [eau du robinet] ; cooling water [eau de refroidissement] ; sewage [eaux usées] ; other (see Description of culture system) [autres (voir le champ Description of culture system].
Supplemental water source [Source d'eau complémentaire] : Mêmes choix que ci-dessus.
Les paramètres physico-chimiques sont : température Temperature,
salinité Salinity, pH, oxygène Oxygen (mg![]() l-1), la saturation en oxygène Oxygen
saturation (%) et l'alcalinité Alkalinity (mg
l-1), la saturation en oxygène Oxygen
saturation (%) et l'alcalinité Alkalinity (mg![]() l-1 CaCO3). Les limites inférieure et
supérieure sont séparées en deux champs, et dans la plupart
des cas, la moyenne des valeurs disponibles ou le milieu de
l'intervalle est calculé dans un troisième.
l-1 CaCO3). Les limites inférieure et
supérieure sont séparées en deux champs, et dans la plupart
des cas, la moyenne des valeurs disponibles ou le milieu de
l'intervalle est calculé dans un troisième.
Description of culture system [Description du système d'élevage] : Contient une description plus détaillée du système de production et de(s) source(s) d'eau.
Main food [Nourriture principale] : Parmi les choix suivants : in-situ production [production in-situ] ; in-situ production plus added feed [production in-situ plus adjonction d'aliment] ; only added feed [adjonction d'aliment seulement].
Feed quantity [Quantité d'aliment] : Indique le poids total d'aliments ajoutés en kg de poids frais ou sec.
% BWD [% PSJ] : Indique une ration journalière en % de poids sec d'aliments rapporté au poids humide de poisson.
Feed quality [Qualité d'aliment] : Indique la teneur protéique de l'aliment en % du poids sec.
Nitrogen [Azote] et Phosphate [Phosphate] en kg·ha-1 ou kg·ha-1·day-1 indiquent la quantité d'azote et de phosphate fournie comme engrais.
Description of nutrient input [Description de la nourriture] : Contient une description détaillée de la nourriture principale, y compris le régime alimentaire, la conversion de la nourriture, etc.
Statut
Bien que le nombre d'espèces de poissons d'élevage reste inférieure à 200, une quantité considérable de données sur l'aquaculture est publiée dans les périodiques scientifiques, les rapports techniques, etc. La progression de la saisie des données est freinée par le manque de standardisation des expérimentations en aquaculture. Ces contraintes tendraient à être minimisées en augmentant les efforts pour décrire et standardiser ces données, et en fournissant un profil d'espèce type (voir ci-dessous).
Le Pond Dynamics/Aquaculture Collaborative Research Support Program (PD/A CRSP) [Programme de soutien aux collaborations de recherche sur la dynamique et l'exploitation piscicole en étang, aux États-Unis] a fait des progrès considérables dans la standardisation des expérimentations en étang (Szyper 1992). Agustin et al. (1993) ont proposé des modèles de format pour la documentation sur les ressources génétiques pour l'aquaculture.
Sources
La table CULTSYS contient plus de 300 enregistrements d'expérience en aquaculture pour environ 15 espèces et souches, extraits entre autres de Hopkins et Cruz (1982), Costa-Pierce et Soemarwoto (1990) et Christensen (1994). La plupart des données n'ont cependant pas été vérifiées. Néamoins, les données de Costa-Pierce et Soemarwoto (1990) ont été entrées sous la direction de Barry Costa-Pierce et les champs ont été examinés par lui. De la même façon, les données de Hopkins et Cruz (1982) ont été vérifiées et analysées par Mark Prein (Prein 1990 ; Prein et al. 1993) et saisies sous son contrôle. D'autres ensembles de données d'aquaculture comme celui de van Dam (1990) et du PD/A CRSP seront inclus dans le futur.
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Fish as food dans la vue BIOLOGY, et sur le bouton Aquaculture dans la fenêtre FISH AS FOOD.
Remerciements
Nous remercions Liza Agustin pour ses contributions à cette table et à une version antérieure de ce chapitre, alors qu'elle était membre de l'équipe FishBase.
Références
Agustin, L.Q., R. Froese, A.E. Eknath et R.S.V. Pullin. 1993. Documentation of genetic resources for aquaculture - the role of FishBase, p. 63-68. In D. Penman, N. Roongratri et B. McAndrew (éds) International Workshop on Genetics in Aquaculture and Fisheries Management. ASEAN-EEC Aquaculture Development and Coordination Programme, Bangkok, Thailand.
Costa-Pierce, B.A. et O. Soemarwoto, Editeurs. 1990. Reservoir fisheries and aquaculture development for resettlement in Indonesia. ICLARM Tech. Rep. 23, 378 p.
Christensen, M.S. 1994. Growth of tinfoil barb, Puntius schwanenfeldii, fed various feeds, including fresh chicken manure, in floating cages. Asian Fish. Sci. 7 : 29-34.
Hopkins, K.D. et E.M. Cruz. 1982. The ICLARM-CLSU integrated animal-fish farming project : final report. ICLARM Tech. Rep. 5, 96 p.
Prein, M. 1990. Multivariate analysis of tilapia growth experiments in ponds : case studies from the Philippines, Israel, Zambia and Peru. Kiel University, Kiel, Germany. Thèse de Doctorat. 125 p.
Prein, M., G. Hulata et D. Pauly, Éditeurs. 1993. Multivariate methods in aquaculture research : case studies of tilapias in experimental and commercial systems. ICLARM Stud. Rev. 20, 221 p.
Szyper, J.P. 1992. A standard format for design and evaluation of pond experiments. Naga, ICLARM Q. 15(4) : 18-20.
van Dam, A.A. 1990. Multiple regression analysis of accumulated data from aquaculture experiments : a rice-fish culture example. Aquacult. Fish. Manage. 21 : 1-15.
Christine Casal et Roger S.V. Pullin
Les expériences d'élevage impliquent souvent plus d'une espèce. Dans ces cas, la table associée CULTSPEC contient la production d'une seule espèce par enregistrement. Dans cette table sont inclus la pratique d'empoissonnement, la période d'élevage, la méthode de écolte, la mortalité pendant la période d'élevage et le rendement brut par cycle de production.
Les champs
Stocking rate [Taux de charge] : Indique la quantité de poissons au début de la période d'élevage. L'unité est précisée parmi les choix : n/m2 ; n/m3 ; n/m3/d ; kg/m3, où n est un nombre d'individus. Cette quantité ne comprend que les individus ou la masse de l'espèce considérée.
Total stocking [charge total] : Indique la biomasse initiale totale de l'espèce considérée en kg.
Stocking weight [Poids de charge] : Indique un poids individuel modal ou typique au moment de l'empoissonnement, en g de poids vivant.
Stocking age [Age de charge] : Indique l'âge moyen en jours des individus au moment de l'empoissonnement. C'est important à cause des poissons à croissance lente, âgés, et qui se reproduisent.
Method used for estimation [Méthode d'estimation] : Indique la méthode utilisée pour l'estimation de la croissance parmi les choix : Ford/Walford ; von Bertalanffy/Beverton ; Gulland et Holt ; régression non-linéaire, ELEFAN ; autres méthodes.
Les paramètres de croissance de la courbe de von Bertalanffy
(L![]() , K) sont indiqués comme paramètres
préférentiels pour la croissance en longueur. Ils sont décrits
plus en détail dans ‹ La table POPGROWTH ›
(ce volume), qui distingue les poissons élevés en captivité
des poissons sauvages (voir aussi Encadré 15 et
Fig. 18).
, K) sont indiqués comme paramètres
préférentiels pour la croissance en longueur. Ils sont décrits
plus en détail dans ‹ La table POPGROWTH ›
(ce volume), qui distingue les poissons élevés en captivité
des poissons sauvages (voir aussi Encadré 15 et
Fig. 18).
Culture period [Période d’élevage] : Indique la durée de production, par exemple des alevins à la taille commerciale, en jours.
Harvesting practice [Méthode de récolte] : Parmi les choix : batch culture [élevage par charges] ; continuous stocking and harvesting [empoissonnement et récolte en continu] ; periodical stocking and harvesting [empoissonnement et récolte périodiques] ; periodical stocking and continuous harvesting [empoissonnement périodique et récolte en continu] ; variable [variable].
Harvesting length [Longueur à la récolte] : Indique la longueur individuelle modale ou typique des poissons à la récolte, en cm.
Harvesting weight [Poids à la récolte] : Indique le poids individuel modal ou typique des poissons à la récolte, en g.
Mature : Indique combien d'individus sont mûrs à la récolte parmi les choix : most [la plupart] ; some [quelques-uns] ; none [aucun].
Mortality (M%) : Indique les pertes rencontrées pendant la période de production en pourcentage, selon l'équation :
![]() ....1)
....1)
où N0 est le nombre initial de poissons, et Nt le nombre à la fin de la période t.
Annual mortality rate (Z) [Taux annuel de mortalité totale] : Selon l'équation :
Z = (ln(No/Nt))/(![]() t
t![]() 365) …2)
365) …2)
où N0 et Nt sont définis ci-dessus, et ![]() t est la période d'élevage, en jours.
t est la période d'élevage, en jours.
Specific growth rate [Taux de croissance spécifique] : En %, selon le calcul :
ln (poids à la récolte - poids à l'empoissonnement)
· 100/![]() t.
t.
Gross yield [Rendement total] : Indique le
rendement total par cycle de production en poids frais, en unité
(ainsi que les rendements ci-dessous) choisie parmi : kg![]() m-2, kg
m-2, kg![]() m-3, kg
m-3, kg![]() m-3j-1, kg
m-3j-1, kg![]() m-2
m-2![]() an-1, kg
an-1, kg![]() m-3
m-3![]() an-1.
an-1.
Net yield [Rendement net] : Est égal au Gross yield [Rendement brut] moins le poids de charge.
Extrapolated yield [Rendement extrapolé] : Indique le rendement total hypothétique qui aurait été obtenu si les conditions étaient restées identiques sur une période de production de 365 jours.
Statut
Un graphique illustre la distinction entre les cycles de croissance des poissons en captivité et des poissons sauvages (voir Fig. 18).
Sources
Voir ‹ Sources › dans ‹ La table CULTSYS › ci-dessus. Noter aussi que la plupart des références utilisées dans cette table concernent des poissons d'eau douce.
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Fish as food dans la vue BIOLOGY, et puis sur le bouton Aquaculture dans la fenêtre FISH AS FOOD et sur le bouton Cultured species dans la vue CULTURE SYSTEM. Double-cliquer sur une des lignes dans la vue LIST OF SPECIES IN POLYCULTURE affiche les informations relatives à l'espèce désignée. Cliquer sur le bouton Production affiche des informations détaillées sur les rendements. Le graphique de la figure 18 est accessible par la fenêtre REPORTS puis par le bouton Population Dynamics de la fenêtre GRAPHS.
Remerciements
Nous remercions Barry Costa-Pierce, Mikkel Christensen, Mark Prein et Anne van Dam d'avoir fourni leurs données pour les diffuser par FishBase et pour leurs suggestions pour l'amélioration des tables CULTSYS et CULTSPEC. Nous remercions Liza Agustin, anciennement membre de l'équipe FishBase, pour ses contributions à cette table et à une version antérieure de ce chapitre.
Roger S.V. Pullin et Christine Casal
Encadré 30. Élevage par sélection du tilapia Nil. Le tilapia du Nil (Oreochromis niloticus) a été l'espèce de tilapia la plus largement élevée depuis 1980. Cependant, Pullin et Capili (1988) ont mis en évidence que très peu d'attention avait été prêtée à l'amélioration génétique de populations d'élevage et que les géniteurs exportés d'Afrique avaient été isolés de très petites populations fondatrices et avaient été probablement mal gérés, entraînant une dérive génétique, un fardeau génétique dû à la consanguinité, et une hybridation introgressive avec d'autres espèces, particulièrement O. mossambicus. Aussi en 1988, un atelier international a-t-il été organisé pour examiner la situation des ressources génétiques du tilapia pour l'aquaculture (Pullin 1988). Il a permis de confirmer la richesse des ressources génétiques de tilapia en Afrique, la diversité génétique limitée des géniteurs de tilapias dans l'aquaculture hors Afrique, et le besoin d'investissements plus importants dans la recherche pour l'amélioration génétique des tilapias. À partir de ces conclusions, en consultation avec les
collègues du AKVAFORSK, Norvège, qui avait été
pionniers dans l'élevage par sélection de saumon
(Gjedrem 1985), du Philippine Bureau of Fisheries and
Aquatic Resources et du Freshwater Aquaculture
Center de Central Luzon State University,
Philippines, l'ICLARM a obtenu du Programme des
Nations-Unies pour le Développement (PNUD) un
financement pour le projet Genetic Improvement of
Farmed Tilapias (GIFT). Avec l'aide de nombreux
collègues et d'institutions en Afrique, Asie et Europe,
quatre nouvelles populations fondatrices sauvages de
tilapias du Nil (Égypte, Ghana, Kenya et Sénégal) et
des populations de quatre souches en usage courant chez
des éleveurs en Asie (Israël, Singapour, Taïwan, et
Thaïlande) ont été rassemblées, après une
quarantaine stricte aux Philippines. Leur performance ont
été comparées dans 11 environnements aquacoles
différents. Le résultat surprenant a été que, à
l'exception de la souche ghanéenne, la croissance des
souches sauvages africaines a égalé voire dépassé
celle des souches d'élevages asiatiques. |
Les membres de l'équipe FishBase ont éprouvé des difficultés à présenter dans un format concis et standardisé, les informations principales sur l'utilisation des espèces de poissons en aquaculture. La littérature disponible manque souvent de données quantitatives et de protocoles, de termes et d'unités standards nécessaires pour des comparaisons simples. D'autre part, cette littérature documente plutôt des expériences de recherche et d'essais de développement plutôt que des opérations aquacoles bien établies. Nous avons aussi constaté que le fait d'assortir et de résumer ces informations pour les saisir dans les tables CULTSPEC et CULTSYS était trop coûteux en temps. Par conséquent, pour augmenter malfré tout la quantité d'informations sur l'aquaculture dans FishBase, nous avons décidé de développer les Profils des Espèces d'Aquaculture : des mini-mémoires de 1 000 mots maximum par espèce, en texte libre mais avec une structure standardisée. Beaucoup de ces informations seront transférées plus tard dans les champs adéquats des diverses tables de FishBase.
Un exemple est donné ci-dessous pour un tilapia, Sarotherodon melanotheron. Nous cherchons des auteurs pour rédiger ces profils pour l'une ou l'autre des espèces d'élevage dans le monde. Le crédit est entièrement attribué à(aux) l'auteur(s). Des illustrations des poissons et des opérations d'élevage, etc., peuvent être incluses. Des suggestions pour améliorer le format de ces profils et pour mettre à jour l'exemple ci-dessous sont aussi les bienvenues.
FishBase 99 contient des profils pour le catla (Catla catla), le chanos (Chanos chanos), le mrigal (Cirrhinus mrigala), le rohu (Labeo rohita) et les tilapias Oreochromis shiranus, Sarotherodon melanotheron et Tilapia rendalli. Des auteurs potentiels pour le profil d'autres espèces devraient vérifier auprès de l'ICLARM que le travail ne soit pas déjà en cours. Il sera plus efficace de nous proposer des espèces dont vous êtes spécialistes, ou dont vous avez au moins de solides connaissances, et pour lesquelles vous disposez de la documentation nécessaire. Nous espérons compléter les profils d'environ 180 espèces pour traiter tous les poissons élevés.
Un exemple de Profil d'Espèce d'Aquaculture :
Scientific Name [Nom scientifique] : Sarotherodon melanotheron Rüppell, 1852.
Pour l'aquaculture, noter qu'il existe cinq sous-espèces avec des traits différents : Sarotherodon m. melanotheron Rüppell, 1852 ; Sarotherodon m. heudelotii (Duméril, 1861) ; Sarotherodon m. leonensis (Thys van den Audenaerde, 1971) ; Sarotherodon m. paludinosus Trewavas, 1983 et Sarotherodon m. nigripinnis (Guichenot 1861) ; pour les descriptions complètes et les synonymies, voir Trewavas (1983).
Common names [Noms communs] : Anglais - black-chinned tilapia (quelque peu équivoque car les motifs de coloration de la tête et du corps varient entre et dans les sous-espèces) ; Français - tilapia ; carpe (aussi équivoque car ce nom est utilisé pour de nombreuses espèces dans de nombreuses familles ; utilisé seulement en Côte d'Ivoire).
History of use [Historique de l'utilisation] :
Utilisé depuis des siècles pour la consommation humaine ;
trouvé dans les eaux saumâtres ouest-africaines et les eaux
douces adjacentes (principalement dans les lagunes, les
estuaires, le cours inférieur des rivières et les lacs et
réservoirs avoisinants), du Sénégal à l'ancien Zaïre ;
poisson d'aquarium populaire, importé pour la première fois en
Europe en 1907 ; son potentiel pour l'aquaculture était
ignoré jusqu'à des tentatives récentes pour adapter à
l'aquaculture extensive les pêches traditionnelles très
productives dans les ‹ acadjas › (paquets de
broussaille dans des lagunes peu profondes, qui attirent les
poissons en leur fournissant un refuge et une nourriture
abondante, surtout du périphyton) dans lesquels de grandes
quantités de cette espèce sont récoltées (7 à 20 t![]() ha-1
ha-1![]() an-1 de poissons
de 20 à 560 g) (Hem et Avit 1996) ; d'autres essais en
étang, en cage et en clôture étaient restés vains, sauf à
produire de petits poissons de 50 g environ (données de
croissance indicatives pour des étangs engraissés et
alimentés, 0,5 à 0,7 g
an-1 de poissons
de 20 à 560 g) (Hem et Avit 1996) ; d'autres essais en
étang, en cage et en clôture étaient restés vains, sauf à
produire de petits poissons de 50 g environ (données de
croissance indicatives pour des étangs engraissés et
alimentés, 0,5 à 0,7 g![]() jour-1 jusqu'à
25 à 35 g avec ralentissement par la suite, production
annuelle de filets, 1,9-3,5 t
jour-1 jusqu'à
25 à 35 g avec ralentissement par la suite, production
annuelle de filets, 1,9-3,5 t![]() ha-1 ;
cages, 0,5 à 0,7 g
ha-1 ;
cages, 0,5 à 0,7 g![]() jour-1 jusqu'à
50 à 60 g et 0,1 à 0,2 g
jour-1 jusqu'à
50 à 60 g et 0,1 à 0,2 g![]() jour-1 ensuite) ; ces essais se sont déroulés
au Bénin, en Côte d'Ivoire et au Nigéria, et ils concernaient Sarotherodon
m. melanotheron ; des travaux très récents en Côte
d'Ivoire (Agnèse 1996 ; Gilles et al. 1998) ont mis
en évidence des croissances beaucoup plus rapides (plus de
2 g
jour-1 ensuite) ; ces essais se sont déroulés
au Bénin, en Côte d'Ivoire et au Nigéria, et ils concernaient Sarotherodon
m. melanotheron ; des travaux très récents en Côte
d'Ivoire (Agnèse 1996 ; Gilles et al. 1998) ont mis
en évidence des croissances beaucoup plus rapides (plus de
2 g![]() jour-1) chez des
poissons provenant des environs de Dakar, Sénégal,
vraisemblablement Sarotherodon m. heudelotii, ou Sarotherodon
m. paludinosus ; des poissons de plus de 200 g ont été
obtenus en six mois, avec un ratio de conversion de nourriture
(poids de nourriture distribuée/ poids frais de poisson
récolté) de 1,7.
jour-1) chez des
poissons provenant des environs de Dakar, Sénégal,
vraisemblablement Sarotherodon m. heudelotii, ou Sarotherodon
m. paludinosus ; des poissons de plus de 200 g ont été
obtenus en six mois, avec un ratio de conversion de nourriture
(poids de nourriture distribuée/ poids frais de poisson
récolté) de 1,7.
Production statistics [Statistiques de production] : aucune encore disponible.
Where farmed [Zones d'élevage] : Afrique de l'Ouest, zone continentale FAO Afrique-01.
Countries [Pays] : Bénin, Côte d'Ivoire, Ghana, Nigeria et Sénégal, et probablement d'autres dans cette région, toujours à petite échelle.
Climate and Environmental Tolerance [Climat et tolérances environnementales] : zone tropicale ; intervalle de température naturelle 17-33°C ; ne se reproduit pas au-dessous de 20-23°C ; tolérance de salinité large, 0-45‰, préfère 10-15‰ ; relativement tolérant aux eaux acides, grandit et se reproduit à pH 3.5-5.2 sur des terrains sulfateux acides (Campbell 1987 ; Trewavas 1983) ; les informations sur les limites létales sont manquantes ; intervalles de tolérance et limites létales varient selon les sous-espèces et les populations.
Current farming methods [Méthodes d'élevage actuelles] : Les méthodes d'incubation sont encore en développement ; se reproduit facilement dans les clôtures, les étangs et les réservoirs ; la production mensuelle d'alevins (1 g) est de 200 000-250 000 dans un système à circuit fermé ; une femelle œuvée pond 200-900 œufs ; la taille à maturité sexuelle est variable selon les populations, de 4,0-4,5 cm LS pour des populations restreintes à 13,4 cm ; le mâle est normalement un incubateur buccal des œufs et alevins ; les alevins se nourrissent principalement de plancton (progressivement plus de zooplancton que de phytoplancton), de détritus et de larves aquatiques ; le régime alimentaire par la suite est omnivore, y compris des détritus (Pauly et al. 1988 ont quantifié son régime détritivore et ont comparé les paramètres de croissance), de plancton, d'invertébrés et de matières végétales, surtout de périphyton ; les alevins acceptent volontiers des aliments à base de son de céréales, de tourteau d'arachide, de farine de poissons et des vitamines (Campbell 1987).
Les méthodes du grossissement sont encore en développement pour les systèmes en étangs, en cages et en clôtures ; les adultes acceptent volontiers des sous-produits agricoles et des aliments en forme de poudre écrasée ou de boulette (Campbell 1987).
Processing and Marketing [Traitement et vente] : Aucune information sommaire disponible ; il est supposé que les produits principaux sont des poissons entiers ou vidés, frais et fumés ou séchés ; les produits à valeur ajoutée, comme les filets, sont attendus si des systèmes intensifs sont développés ; les grands poissons (de plus de 350 g) et les produits à valeur ajoutée pourraient entrer sur les marchés mondiaux de tilapias ; les plus petits poissons (environ 50 g) sont importants dans les marchés domestiques.
Likely Future Trends [Tendances futures vraisemblables] : Pourrait devenir important dans l'aquaculture en Afrique de l'Ouest, si les systèmes en développement tiennent leurs promesses initiales ; intérêt pour l'aquaculture en eau saumâtre dans d'autres régions à cause de sa large tolérance vis-à-vis de la salinité ; pour cela, des estimations adéquates des impacts possibles sur l'environnement sont essentielles avant de quelconques introductions, étant donné les expériences passées négatives avec un autre tilapia (Oreochromis mossambicus) ; des études précises des caractéristiques des différentes sous-espèces et populations, et leur conservation pour l'utilisation dans des programmes d'élevage sont nécessaires.
Comment y arriver
Cliquer sur le bouton Importance dans la vue SPECIES et sur le bouton Profile dans la fenêtre IMPORTANCE.
Références
Agnèse, J.F. 1996. La recherche au service du développement : l'exemple du programme GENETICS. Bull. CE Coopération Pêche 9(3) : 16-17. [version anglaise disponible en p. 15-16 de la même série].
Campbell, D. 1987. A review of the culture of Sarotherodon melanotheron in West Africa. UNDP/FAO African Regional Aquaculture Centre, Aluu, Port Harcourt, Nigeria. Working Paper ARAC/87/WP/5, 20 p.
Gilles, S., J.B. Amon-Kothias et J.-F. Agnèse. 1998. Comparison of brackishwater growth performances of Sarotherodon melanotheron (Cichlidae) from three West African population, p. 199-210. In J.-F. Agnèse (éd.) Genetics and aquaculture in Africa. ORSTOM, Africa.
Hem, S. et J.L.B. Avit. 1996. Acadja-enclos : un système d'exploitation piscicole extensive en Côte d'Ivoire, p. 48-55. In R.S.V. Pullin, J. Lazard, M. Legendre, J.B. Amon-Kothias et D. Pauly (éds). Le Troisième Symposium International sur le Tilapia en Aquaculture. ICLARM Conf. Proc. 41, 630 p. [Disponible en p. 45-53 dans la version anglaise].
Pauly, D., J. Moreau et M.L. Palomares. 1988. Detritus and energy consumption and conversion efficiency of Sarotherodon melanotheron (Cichlidae) in a West African lagoon. J. Appl. Ichthyol. 4 : 190-193.
Trewavas, E. 1983. Tilapiine fishes of the genera Sarotherodon, Oreochromis and Danakilia. British Museum (Natural History), London. 583 p.
Roger S.V. Pullin
Les maladies constituent un problème majeur en aquaculture intensive, dans le commerce aquariophile, et dans les baies, lagunes et eaux continentales polluées. Les ordinateurs peuvent aider à diagnostiquer des maladies des poissons, d'une manière similaire à leur utilisation pour l'Identification Assistée par Ordinateur des larves (voir ‹ La table LARVAE) ›, ce volume).
Sources
La table DISREF a été originellement développée par Imke Achenbach et Rainer Froese (Achenbach 1990 ; Achenbach et Froese 1990). Elle contient 314 descriptions de maladies ou stades de maladies, extraites de plus de 200 références. Environ 150 symptômes macroscopiques ont été identifiés et peuvent être utilisés comme des critères diagnostiques. Achenbach et Froese ont montré que les informations déjà compilées peuvent être utilisées pour diagnostiquer des maladies des espèces marines d'aquaculture de l'hémisphère nord (Achenbach et Froese 1990).
Heino Möller et Kertin Anders nous ont récemment donné l'autorisation d'utiliser les illustrations de leurs livres et leur collection de diapositives (Möller et Anders 1983, 1986, 1989), augmentant ainsi le nombre des illustrations des maladies de 3 à 267 (voir ‹ Pictures ›).
Statut
Deux experts, Toshihiko Matsusato et Brian Jones, ont vérifié les informations déjà compilées et nous avons intégré leurs suggestions et leurs corrections. Cependant, nous estimons que la table est encore à un stade de prototype et ne devrait pas être utilisée en routine. Nous apprécierions plutôt qu'une institution qui travaille sur les maladies des poissons prenne la responsabilité de cette table et de la suivante, soit complètement soit seulement pour certains groupes de maladie, et soumettent leurs tables à une vérification consciencieuse et à un développement supplémentaire.
La table DISEASES contient des signalements de maladies référencés. Pour chaque cas, sont indiqués : Species [Espèce] (l'espèce de poisson affectée) ; Disease [Maladie], Country [Pays] et Locality [Localité], Year [Année], Prevalence [Prévalence], Intensity [Intensité], Mortality [Mortalité] (due à la maladie), et des informations additionnelles. Elle contient 218 enregistrements de 148 maladies signalées pour 38 espèces.
Comme mentionné pour la table DISREF ci-dessus, cette table est encore un prototype et ne devrait pas être utilisée en routine. Pour plus d'informations, veuillez contacter l'équipe FishBase.
Comment y arriver
Cliquer sur le bouton Biology dans la vue SPECIES, puis sur le bouton Morphology and physiology dans la vue BIOLOGY, puis sur le bouton Diseases dans la fenêtre MORPHOLOGY & PHYSIOLOGY pour la table DISEASES et sur le bouton More information dans la vue DISEASES table DISREF.
Remerciements
Nous remercions Heino Möller et Kerstin Anders pour avoir autorisé l'intégration de leurs illustrations de maladies dans FishBase.
Références
Achenbach, I. 1990. Aufbau und Entwicklung eines rechnergestützten Informationssystems zur Identifikation von Fischkrankheiten. Christian-Albrechts-Universität, Kiel. 58 p. Thèse de Master.
Achenbach, I. et R. Froese 1990. Presentation of a database system for information on and diagnosis of fish diseases. ICES C.M.1990/F : 72, 13 p.
Möller, H. et K. Anders. 1983. Krankheiten und Parasiten der Meeresfische. Verlag Möller, Kiel. 258 p.
Möller, H. et K. Anders. 1986. Diseases and parasites of marine fishes. Verlag Möller, Kiel. 365 p.
Möller, H. et K. Anders. 1989. Krankheiten und Parasiten der Meeresfische, 100 dias. [Les maladies et les parasites des poissons marins : 100 diapositives]. Verlag H. Möller, Kiel, Germany.
Rainer Froese